ちょ〜高効率DNAクローニング法
Ultra-Low Background DNA Cloning System
詳細な実験手順はbio-protocol.orgの中のこのページにあります。(ごめんなさい、英語です。)
過去の方法を改良してDNAクローニング効率がほぼ100%の方法を開発しました。詳細は上記の論文をご覧下さい。以下は、この方法の日本語解説です。(すみませんが、図は英語のままです。)研究材料の入手方法については、ここをご覧下さい。論文に使った図は、クリエイティブ・コモンズ(Creative Commons)ライセンスのもとで配布が可能なので、論文で使った図をそのまま使いながら以下に説明します。
1.効率が良いDNAクローニング法に、相同組換えによるパン酵母in vivoクローニング法があります。この方法の詳細については、我々の「過去の方法のHP」や岡山大学の守屋先生のHPをご覧下さい。今回、私たちは、この方法を改良して、大腸菌プラスミドへのクローニング効率がほぼ100%の方法を開発しました。以下では、相同組換えによるin vivoクローニングがわかっているという前提で説明しますが、まあ、相同組換えを簡単に言うと同じ配列同士がくっつく反応です。クローニングしたいDNA断片にプラスミドと相同な短いDNA配列を持たせてあげると、クローニングしたいDNA断片がプラスミドにくっつき、その結果、DNA断片がプラスミドにクローニングできます。パン酵母では、この相同組換えが高効率でおこります。ところで、この研究は、一見、酵母の研究のように思えますが、実は、大腸菌のプラスミドの研究である点にご注意ください。つまり、大腸菌プラスミドへのDNAクローニング効率ほぼ100%を達成したのです。この方法の特長は大腸菌と酵母の二つの複製開始点を使って、クローニング副産物をほぼゼロにしたことです。以下の説明を見ながら理解して頂けたら、ありがたいのですが、論理は複雑です。
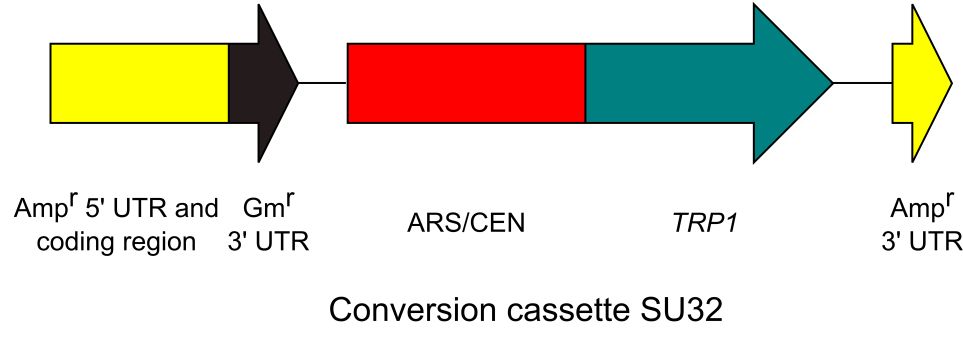
2.この研究で、下図のように変換カセットSU32というDNA断片を作成しました。プラスミドに良く乗っかっているアンピシリン抵抗性遺伝子を終止コドンのところで、ぶった切って、その間に、ゲンタマイシン抵抗性遺伝子のしっぽのところのパリンドロームの部分、酵母のARS/CEN(まあ、複製と分配に必要な配列)と栄養要求性選択マーカーTRP1を挿入したものです。

3.ちなみにこれを作成するときに鋳型は以下のようなものなんですが、大腸菌複製開始点をもっていません。大腸菌複製開始点をもつプラスミドを鋳型にしちゃうと、その鋳型そのものが、大腸菌の中でも酵母の中でも生き残って、クローニング副産物になっちゃいます。でも、この鋳型をたくさん調製することができないのが難点ですが、そこはPCRという文明の利器でクリアできるわけです。
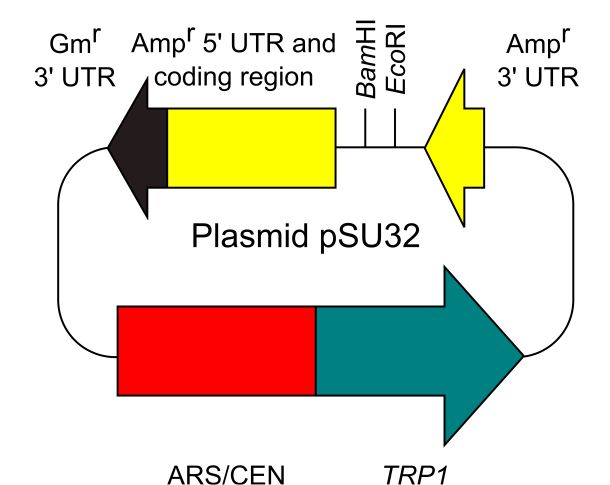
4.下図のように、このカセットを使うと、アンピシリン抵抗性遺伝子をもつプラスミドベクターを大腸菌・酵母シャトルベクターに酵母細胞の中で変換できます。アンピシリン抵抗性遺伝子の部分で、相同組換えがおこるわけです。ちなみに、アンピシリン抵抗性遺伝子をもつプラスミドベクターが、この業界では一番頻繁に使われていることはご存じかと思います。
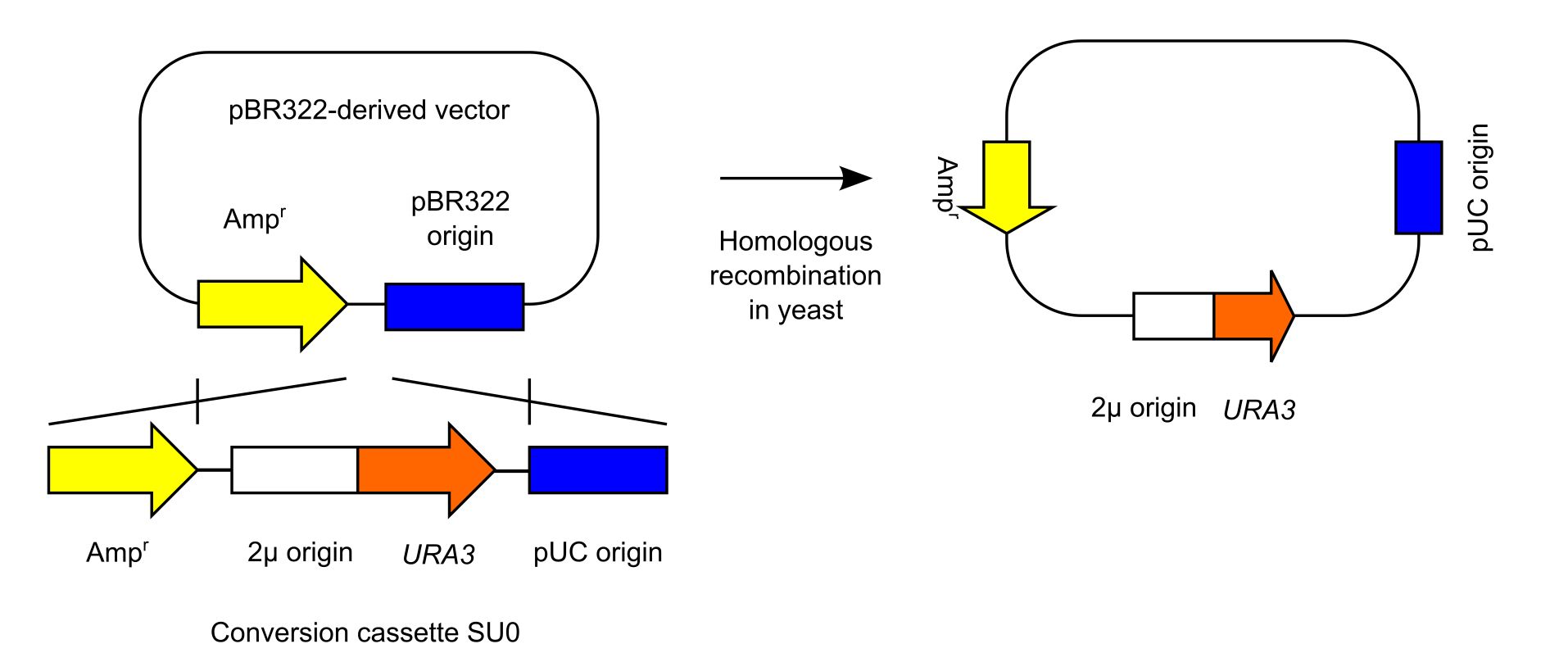
5.この方法は、下図に示した過去の方法と似ています。以下の図で示した過去の方法では、アンピシリン抵抗性遺伝子の部分とColE1起点の部分の二箇所で、相同組換えがおこり、大腸菌・酵母シャトルベクターに変換されたわけです。一方、新しい方法では相同組換えが起こる場所が、アンピシリン抵抗性遺伝子のみになりました。これには、相同組換えをおこさせる領域をアンピシリン抵抗性遺伝子の部分だけにして、従来法よりも汎用性を高めるという意味があります。しかし、もっと重要な意味、すなわち、クローニング副産物の排除という意味があります。過去の方法ですと、非相同末端結合という細胞内の反応で変換カセットSU0が環状化したもの(副産物)は、大腸菌と酵母の複製開始点を二つ持つことになります。この副産物は、大腸菌の中でも酵母の中でも生き残ってしまいます。一方、上記の変換カセットSU32が環状化したものは、酵母の中では生き残っちゃいますが大腸菌の中では生き残れません。
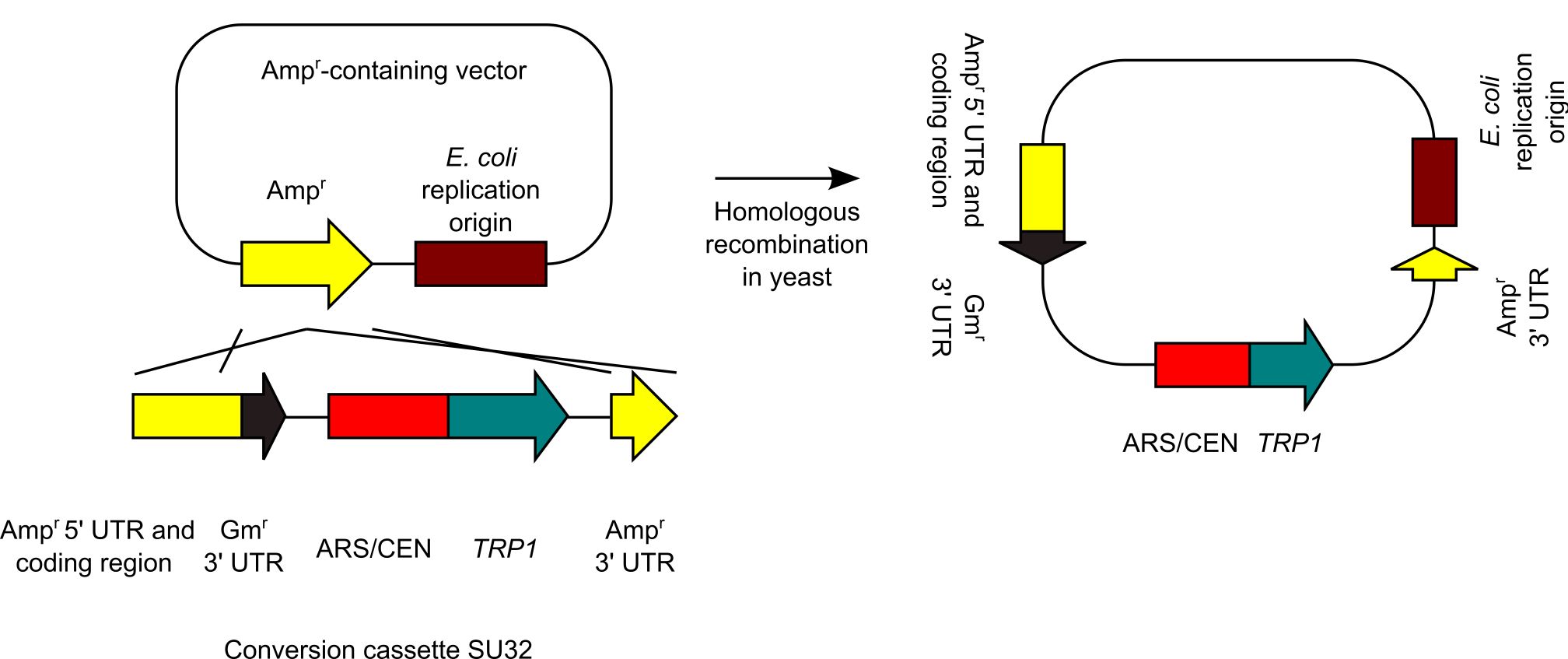
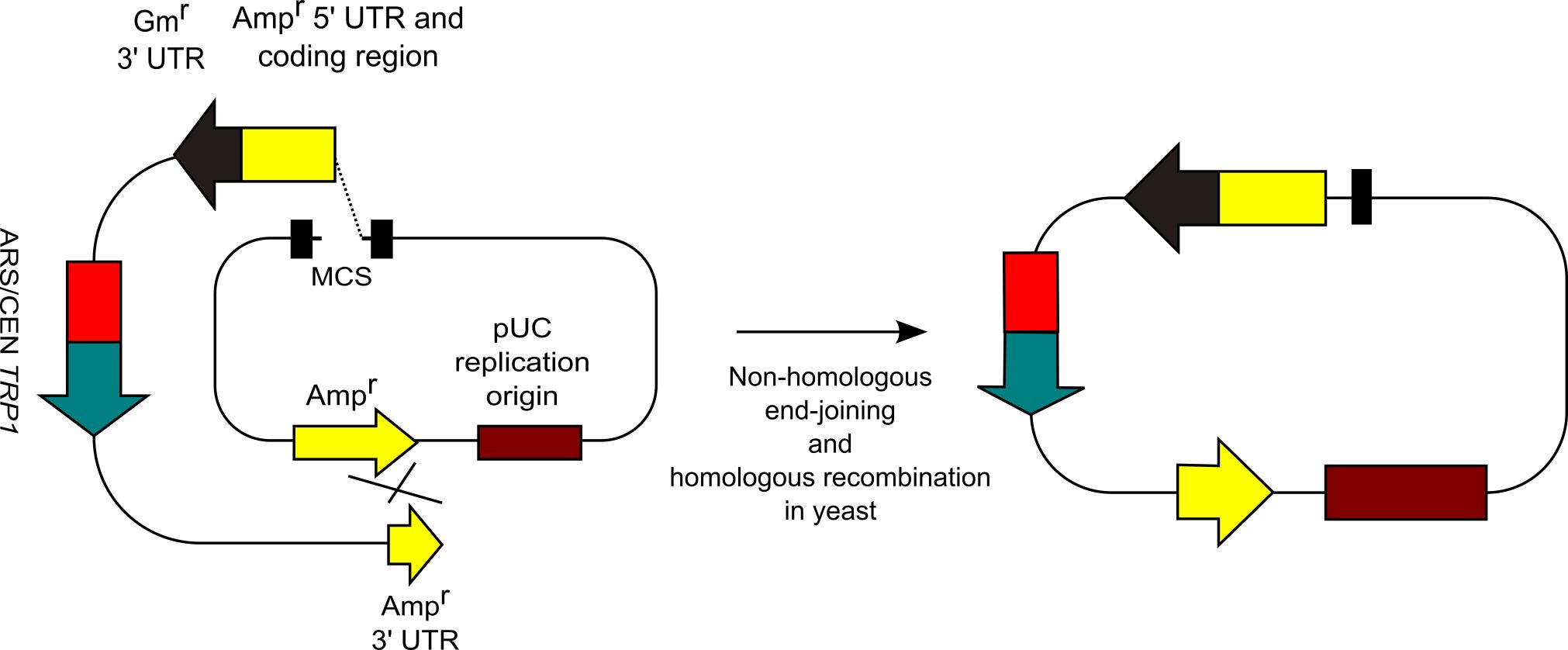
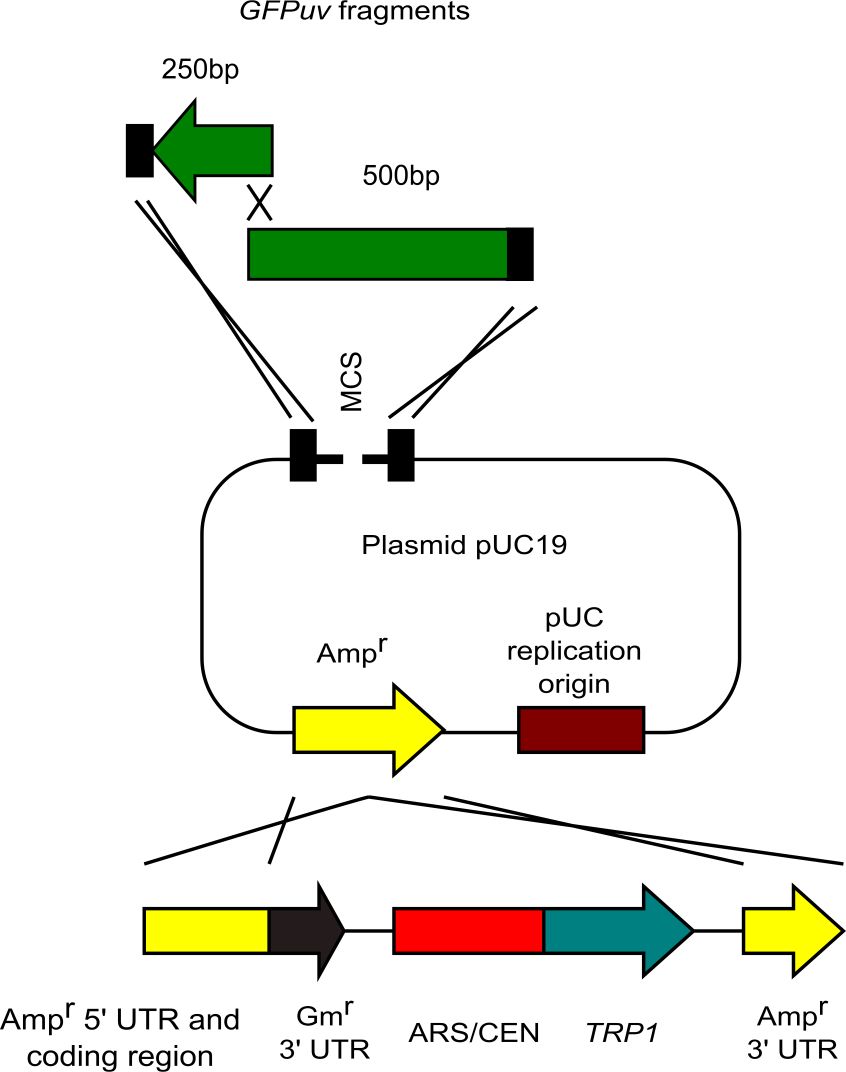
6.過去の方法では、5.の方法で大腸菌プラスミドを大腸菌・酵母シャトルベクターに変換すると同時に、クローニングしたいDNA断片を挿入するということをしました。この考え方は、下図の新しい方法でも同じで、大腸菌プラスミドを大腸菌・酵母シャトルベクターに変換すると同時に、クローニングしたいDNA断片を挿入しました。実際の実験ではGFPのDNA断片を大腸菌プラスミドpUC19に入れています。実際の実験を説明しましょう。変換カセットSU32、クローニングしたいDNA断片が交叉する二箇所の中間地点をぶった切ったpUC19(この切断は必須)、クローニングしたいDNA断片であるGFPのDNA断片を酵母に形質転換しました。この図では、MCSでぶった切っていますが、別にMCSで切ることは重要ではありません。交叉する二箇所の間を切ることが重要です。この柔軟性も、パン酵母in vivoクローニング法の良い点です。この際、酵母の中でいろいろな反応がおこりますが、切れ残ったpUC19 (Undigested pUC19)、非相同末端結合で環状化したpUC19 (Circularized pUC19)、相同組換えでGFPがpUC19に挿入されたのみのもの (pUC19 carrying GFP only)は、酵母複製開始点がないため排除されます。さて、酵母の中では、何が残るのでしょうか?もちろん、目的産物(Final Product)も残るのですが、他に非相同末端結合で環状化した変換カセット(Circularized coversion casette)、切れ残ったpUC19 に変換カセットが挿入されたもの(Undigested pUC19 carrying conversion cassette only)、非相同末端結合で環状化したpUC19変換カセットが挿入されたもの (Circularized pUC19 carrying conversion cassette only)が、とりあえず、残るものとして思いつきます。このうちの環状化した変換カセット(Circularized coversion casette)がメジャーな副産物と考えられますが、これは大腸菌の中で生き残ることができません。要は、複製開始点を利用して、主要な副産物を除去できるわけです。マイナーな副産物の除去方法(下図のOptionalの部分)については後で解説します。
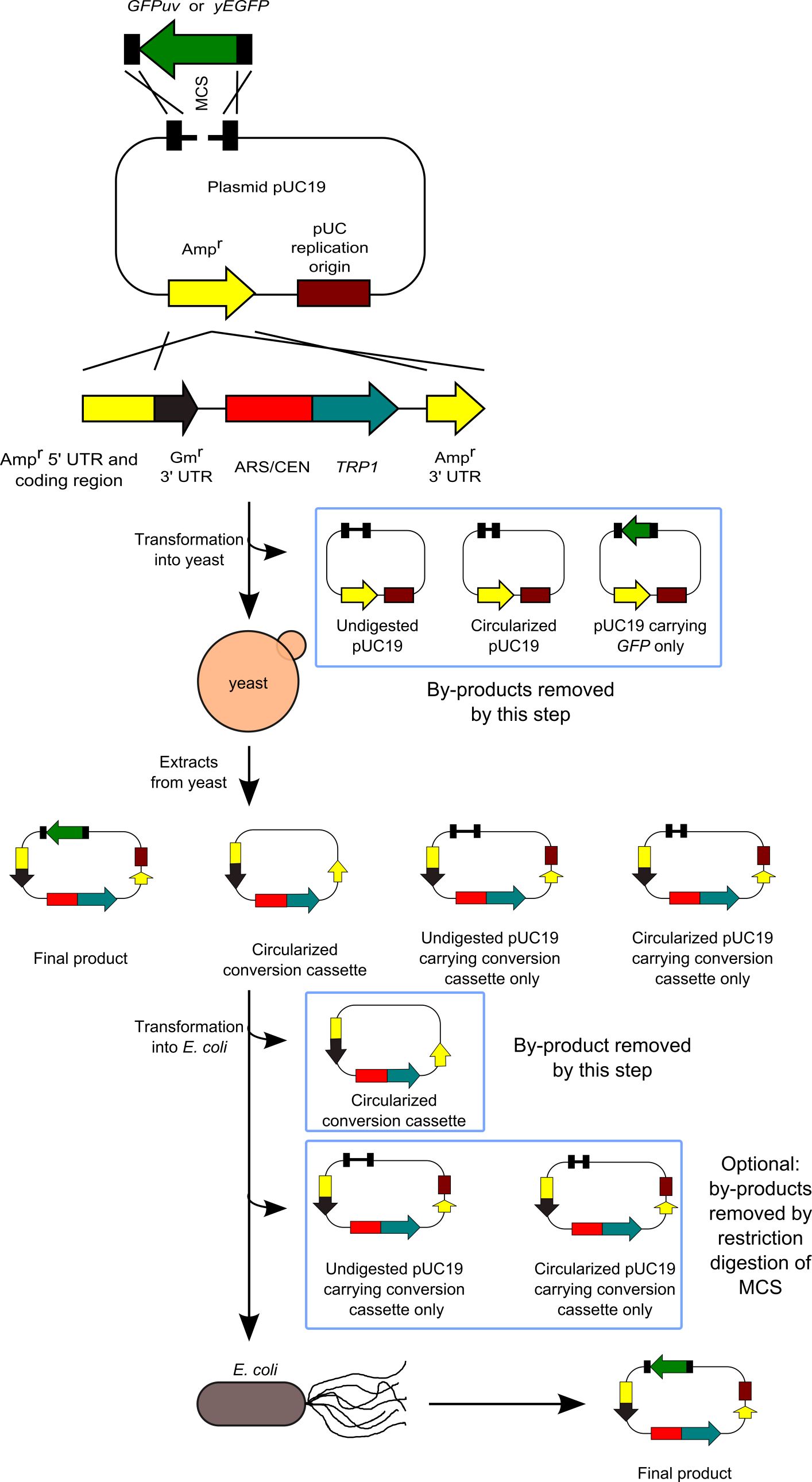
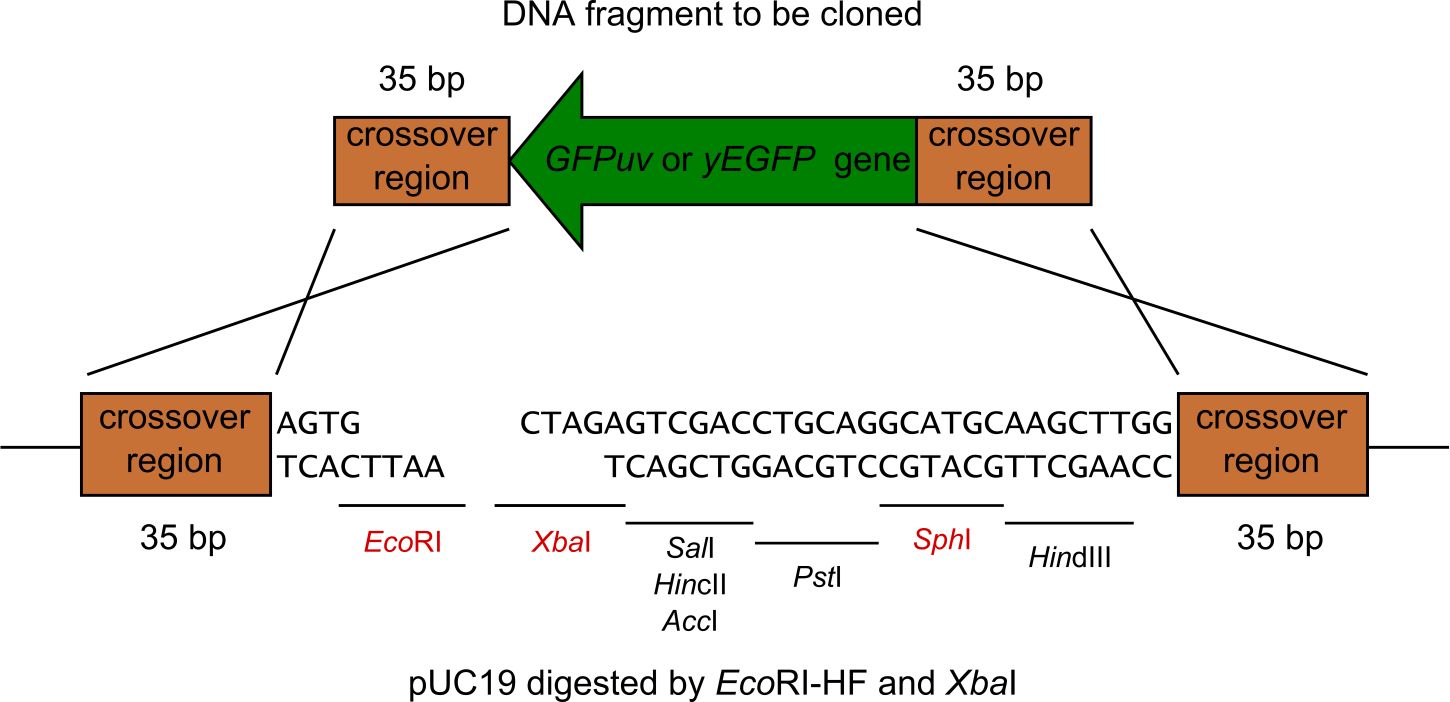
7.下図は、6.の図のGFPを挿入する部分の詳細図です。相同組換えがどこでおこるのかがわかると思います。この後の説明を理解するのに重要なのですが、プラスミドの端っこで組み換え反応を起こさないように敢えて設計しています。
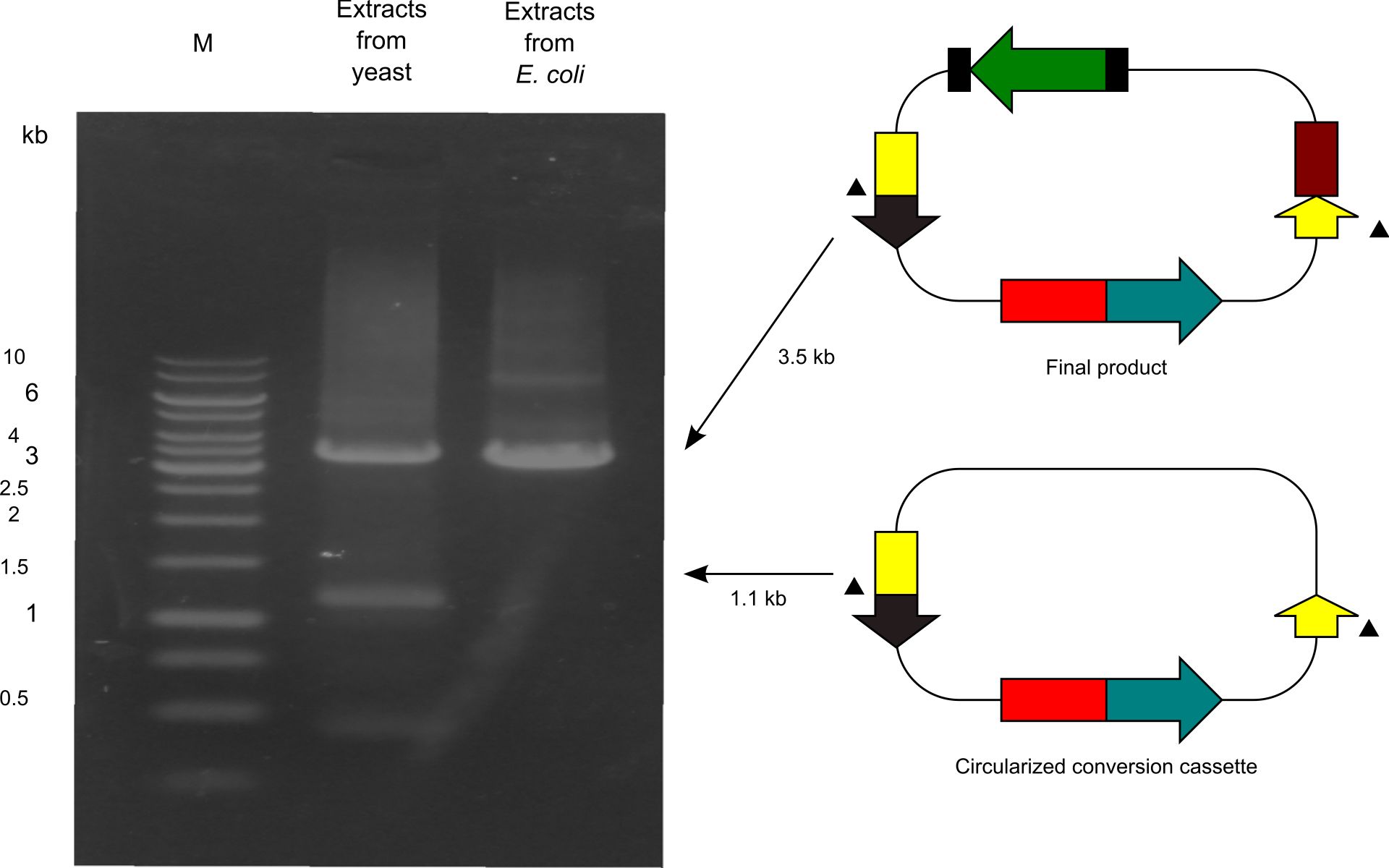
8.下図では、酵母からの抽出液をPCRで解析した結果を示しています。目的産物(Final Product)と環状化した変換カセット(Circularized coversion casette)が検出されました。環状化した変換カセット(Circularized coversion casette)がメジャーな副産物という予測が当たったわけです。しかし、それ以外のマイナーな副産物はPCRでは検出されませんでした。また、当たり前ですが、この図から、大腸菌からの抽出液に環状化した変換カセット(Circularized coversion casette)が検出されないこともわかります。
9.下表は、6.で示した実験(ただし、Optionalの部分はしていない)の結果をを示しています。三回実験しました。(本当は、試行錯誤の過程でもっともっと実験をしていますが。)結果はまあまあでした。
|
|
Digestion
by SphI |
Insert |
Number
of E. coli transformants containing
the insert(s)/number of tested E. coli
transformants (percentage) |
|
Experiment
1 |
No |
GFPuv |
1895/1904
(99.5%) |
|
Experiment 2 |
No |
GFPuv |
2829/2839
(99.6%) |
|
Experiment 3 |
No |
GFPuv |
984/1004
(98.0%) |
10.そこで、まあまあの結果であった原因を探りました。具体的には、副産物が何であるかを探りました。すると、切れ残ったpUC19 に変換カセットが挿入されたもの(Undigested pUC19 carrying conversion cassette only)が検出されました。これはマイナーな副産物であろうとあらかじめ予測していたものです。もう一つ下図のマイナーな副産物がありました。これはあらかじめ予測すべきだったんですが、実験結果を見て気がついたわけです。相同組換えと非相同末端結合の組み合わせで生じる副産物です。(後藤くんがアンピシリン抵抗性遺伝子の頭の配列を記憶していてくれたおかげで、直ぐに正体に気がついたことは幸運でした。)
11.これら副産物たちを、除くことができます。検出された副産物である「切れ残ったpUC19 に変換カセットが挿入されたもの(Undigested pUC19 carrying conversion cassette only)」および検出されなかった副産物である「非相同末端結合で環状化したpUC19変換カセットが挿入されたもの (Circularized pUC19 carrying conversion cassette only)」は、6.の図のMCSの部分を切断すれば大腸菌に形質転換したときに排除されます。これが6.の図のOptionalの部分の理屈です。7.の図で説明すると、制限酵素SphIなどで切断すると良いわけです。ところで、非相同末端結合の結果、10.で説明した副産物に、MCSのSalIサイトよりも右の部分が残ります。すなわち、この副産物も制限酵素SphIで切断すると排除できるのです。そこで、この考え方に基づいて実験をしました。下表は、6.で示した実験(ただし、Optionalの部分を含む)の結果を示しています。三回実験しました。見事な結果でした。
|
|
Digestion
by SphI |
Insert |
Number
of E. coli transformants containing
the insert(s)/number of tested E. coli
transformants (percentage) |
|
Experiment 4 |
Yes |
GFPuv |
2044/2047
(99.9%) |
|
Experiment 5 |
Yes |
GFPuv |
2028/2032
(99.8%) |
|
Experiment 6 |
Yes |
GFPuv |
2807/2813
(99.8%) |
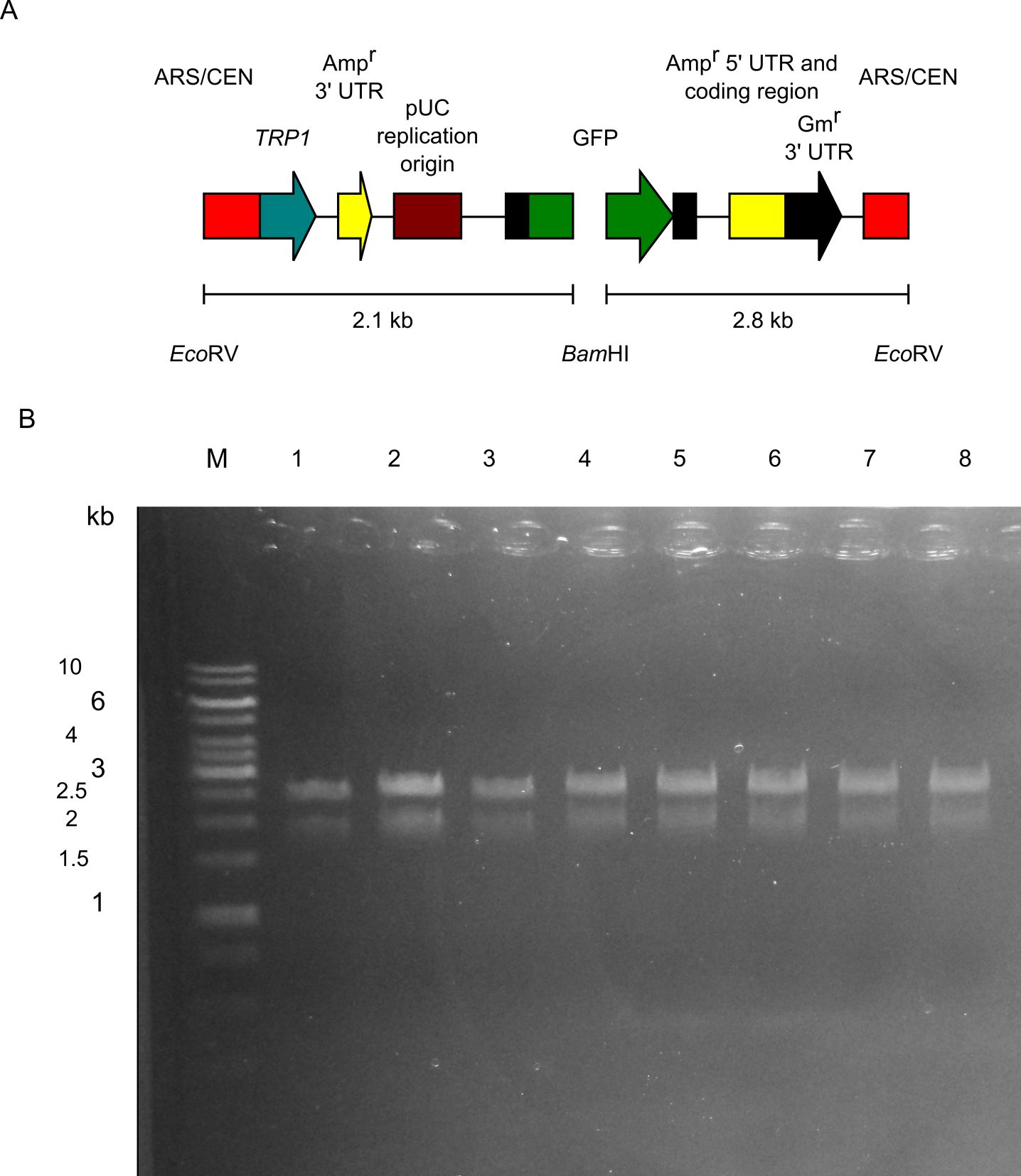
12.下図は緑に光ったコロニーをつまんで、そのプラスミドを解析した図です。予測した制限酵素パターン(下図のA)とプラスミドを解析の結果(下図のB)が一致しました。8.の図はエチジウムブロマイドで染色したものですが、下図はGelRedで染色したもので、多少見え方が違います。
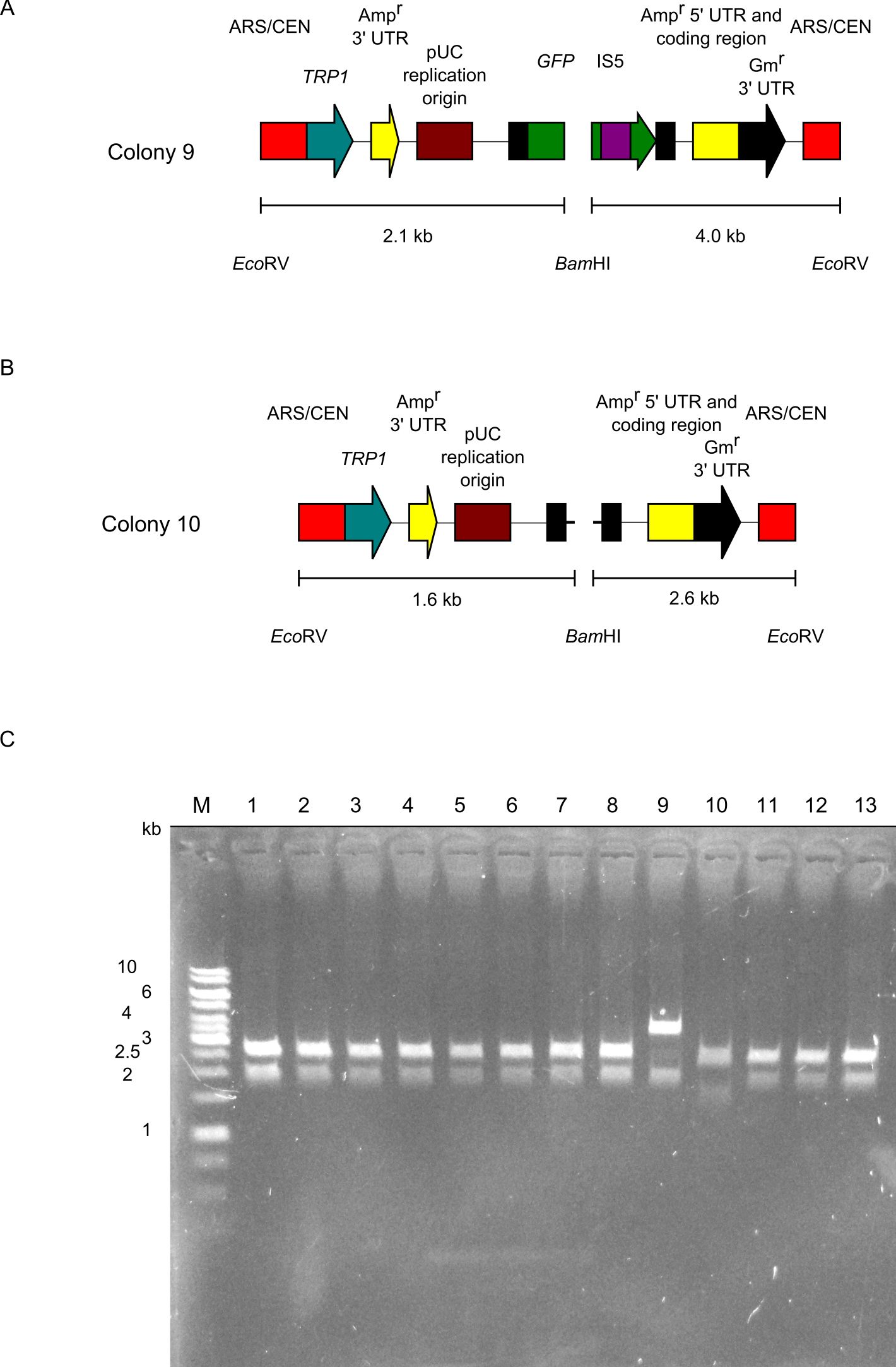
13.下図は緑に光らなかった13個のコロニー(Experiments 4-6の白コロニー全部)をつまんで、そのプラスミドを解析した図(下図のC)です。また、その解析結果を表にまとめたものも下記に示しました。コロニー9と10を除いた11個のコロニーを解析した結果は、11.の図のAのパターンと一致しています。この11個のうち、コロニー8を除いた10コロニーでは、プライマー部にエラーがおこっていました。今回の実験では、PAGE精製したプライマーを用いましたが、プライマー合成の際のエラーを完全に排除することはできないのです。コロニー8では、変異がないGFP遺伝子が挿入されていました。このプラスミドを大腸菌に入れ直すと、緑に光りますので、実験のどこかで、宿主の大腸菌染色体に変異が入ったため白コロニーになったのだと考えられます。これは非常に稀なイベントです。次に、コロニー9と10についての解析結果を説明します。まず、コロニー10は切れ残ったpUC19 に変換カセットが挿入されたもの(Undigested pUC19 carrying conversion cassette only)でした(下図のB)。EcoRI、XbaI、SphIによる制限酵素攻撃をエスケープしたものです。なんと、コロニー9では転移因子のIS5が挿入されていました(下図のA)。IS5の標的サイトは5′-CTAGなのですが、見事に標的サイトに挿入されていました。これは非常に稀なイベントです。言い方を変えると、このようなレアなイベントを検出できるくらい、今回開発したDNAクローニング法は効率が高いのです。
|
Colony
number |
Experiments |
Reasons
for not expressing GFPuv |
|
1 |
Experiment
4 |
Error
at the sites targeted by the PCR primer (717-719 ATA in GFPuv was changed to TTC) |
|
2 |
Experiment 4 |
Error
at the sites targeted by the PCR primer (G was inserted after 16G in GFPuv) |
|
3 |
Experiment 4 |
Error
at the sites targeted by the PCR primer (4G in GFPuv was deleted) |
|
4 |
Experiment 5 |
Error
at the sites targeted by the PCR primer (C was inserted after 5C in GFPuv) |
|
5 |
Experiment 5 |
Error
at the sites targeted by the PCR primer (G was inserted after 16G in GFPuv) |
|
6 |
Experiment 5 |
Error
at the sites targeted by the PCR primer (7A in GFPuv was deleted) |
|
7 |
Experiment 5 |
Error
at the sites targeted by the PCR primer (G was inserted after 4G in GFPuv) |
|
8 |
Experiment 6 |
Uncharacterized
mutation(s) in E. coli chromosome |
|
9 |
Experiment 6 |
IS5
was inserted after 538G in GFPuv
and before 535C in GFPuv |
|
10 |
Experiment 6 |
Undigested
pUC19 carrying conversion cassette only (Escape from the restriction
digestions) |
|
11 |
Experiment 6 |
Error
at the sites targeted by the PCR primer (709C in GFPuv was deleted) |
|
12 |
Experiment 6 |
Error
at the sites targeted by the PCR primer (7A in GFPuv was deleted) |
|
13 |
Experiment 6 |
Error
at the sites targeted by the PCR primer (709C in GFPuv was deleted) |
|
|
Digestion
by SphI |
Insert |
Number
of E. coli transformants containing
the insert(s)/number of tested E. coli
transformants (percentage) |
|
Experiment 7 |
Yes |
yEGFP |
1590/1590
(100%) |
|
Experiment 8 |
Yes |
split GFPuv |
624/628
(99.4%) |
15.今後は、DNAクローニング効率がほぼ100%であることを生かした研究を展開します。今後の研究内容は秘密。
16.この研究は後藤健太くんという学生と二人で行ったものです。