この方法は古い方法です。新しい方法については、こちらをご覧下さい。
新しい遺伝子組換え技術の開発
酵母相同組換え法とその活用
-特に「高効率組換えDNA分子構築法」について-
1.なぜ「高効率組換えDNA分子構築法」が必要か?
DNA断片をベクターにクローニングすることはバイオ実験の基本中の基本です。しかし、基本中の基本であるにもかかわらず、この実験の効率は決して良いものでありません。特に、クローニングしようとしているDNA断片が2~3kbを超える場合や、ベクターが10kbを超える場合に顕著に難しくなります。ある計算によれば、この実験に失敗して、世界中で五分に一度、学生が先生に怒られています。
そこで、これを解決する簡単な実験方法を開発しました。
これは私の最初の卒論生の飯笹英一くんが卒業研究中に開発したものです。
2.「高効率組換えDNA分子構築法」の原理は?
電離放射線等で、DNA二本鎖がどちらも切断されてしまうことがおこりますが、これは細胞にとって極めて危険なことです。そこで、細胞は、1)相同組換え修復と2)非相同的末端連結によって、DNA二本鎖切断を修復します。酵母では1)の相同組換え修復が高頻度で作動します。この性質はプラスミド構築にこれまでも利用されていました。具体的には、制限酵素で切断したベクターおよびそのベクターとの相同配列をPCRで付加させたDNA断片を酵母に同時に形質転換すると、相同配列が組換わり、DNA断片がベクターに組込まれるという方法です。酵母相同組換え法は制限酵素サイトに依存せず、大きなDNA断片や複数のDNA断片を容易にクローニングすることが出来ます。しかし、多くのベクターは酵母の複製開始点を持たないため、この方法は酵母のベクターにDNA断片をクローニングする時にしか用いることがこれまでは出来ませんでした。
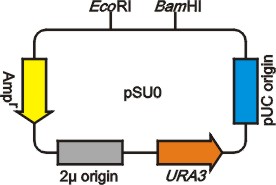
そこで、今回私たちは、この酵母相同組換えによるクローニングを酵母のベクター以外に拡張しました。ほとんどのベクターはpBR322というプラスミドを元に作られているため、アンピシリン耐性遺伝子(Ampr)と複製開始点pBR322
origin (またはその派生物であるpUC origin)の配列を直列に持っています。私たちが作製したヘルパープラスミドpSU0はこの2つの配列の間に酵母の複製開始点2μ
originおよび、URA3遺伝子を持っています(下図)。

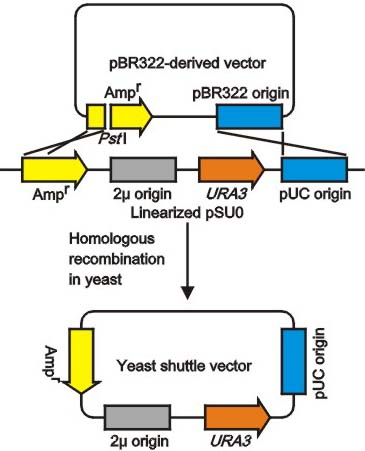
そのため、切断したpSU0とpBR322由来ベクターを同時に酵母に形質転換すると、相同組換えにより、様々なpBR322由来ベクターを酵母シャトルベクターに変換することができます(下図)。
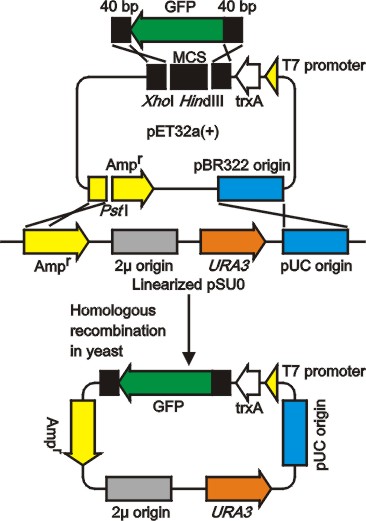
さらに、1)切断したpSU0、2)pBR322由来ベクター、3)PCRで相同配列を付加したDNA断片の三つ(DNA断片の数で4つ)を同時に酵母に形質転換することにより、相同配列が組換わり、1回の形質転換で、pBR322由来ベクターが酵母シャトルベクターに変換されると同時にDNA断片がそのベクターに組込まれます(下図)。クローニング効率ですが、下のモデル実験の場合で、DNA断片の比によらず80~90%でした。また、幾つか他のDNA断片を試しましたが、概ね、この方法のクローニング効率は70~100%でした。
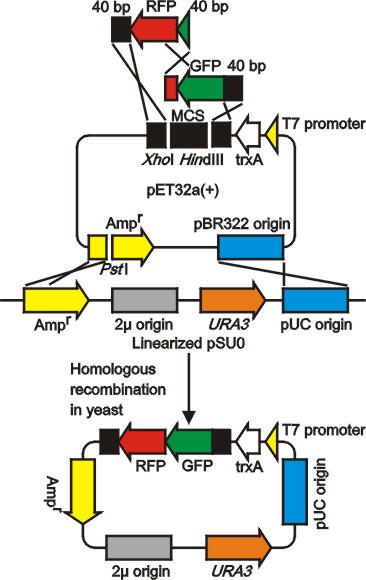
また、言葉で説明するよりも下図を見てもらった方がわかりやすいですが、この方法で多断片連結も可能です。下のモデル実験の場合で、クローニング効率は65%でした。この多断片連結ですが、DNA断片によってはクローニング効率が100%近いこともあります。また、断片の数が増えると効率が下がりますが、6断片を一カ所で連結することに成功しています。このときのクローニング効率は約10%でした。
なお、この方法の詳しい実験結果や細かい実験方法については下記の論文を参照ください。
3.GATEWAY法との比較
酵母相同組換え法もGATEWAY法も共に、網羅的2ハイブリッドのようなハイスループットな実験に用いられた実績があります。したがって、ハイスループットな実験にはどちらの方法も適しています。
GATEWAY法は、ベクター上の決められた位置に、決められた向きで、DNA断片をクローニングする際には便利です。しかし、ベクター上の好みの位置に、好みの向きで、DNA断片をクローニングすることはできません。一方、私たちの「高効率組換えDNA分子構築法」はベクター上の好みの位置に、好みの向きで、DNA断片をクローニングすることができます。また、多断片連結を繰り返し行うこともできます。すなわち、私たちの開発した方法は、網羅的2ハイブリッドのようなハイスループットな実験だけでなく、ふだん行っているいつもの実験にも有利です。目的遺伝子、プロモーター、ターミネーター、レポーター遺伝子、タグ用DNA断片などを好きなように組み合わせてプラスミド構築を行うことが可能です。
4.ヘルパープラスミドpSU0、pSU7、pSU22、およびpSU23について
これらの入手法はこちらをご覧ください。
5.このシステムを利用している研究者からのメール
某大学(佐賀大学ではない)の先生より
「いただいた酵母システムで、我々が研究している癌抑制遺伝子の哺乳類細胞発現プラスミド(pCR3.1)からexon17だけをdeleteしたmutantを作製することができました。このシステムを使えば制限酵素サイトをあまり気にせず、自由自在にmutantを作製できそうです。今レトロウイルスベクターでも同じことをしています。高分子のチャネル遺伝子の研究でも活用しようと考えています。ありがとうございました。」
6.この方法の欠点
概ね、この方法のクローニング効率は70~100%なのですが、まれに相同組換えされることを嫌がるDNA断片があります。でも、どうしてなのかわかりません。また、クローニングする部分の前と後に同じ向きで同じ配列があるとそこで組換えがおこります。
7.この方法に関する質問
この方法に関する質問は、永野nagano@cc.saga-u.ac.jpまでお寄せください。なお、細胞周期に関する細かい質問(例えば、「論文のFig. 2Aで、相同組換え修復と非相同的末端連結が同時に起こるイベントがあったと主張しているが、この二つのイベントがおこる細胞周期における時期は異なるのでは?」といった質問)はわかりません。
8.今後の展開について
酵母を直接研究対象としない私たちの視点で酵母相同組換え法を眺めると、酵母を直接研究対象とする研究者が思いつかない様々な研究の展開が見えてきます。そこで、酵母相同組換え法を用いていくつか研究を展開していく予定です。また、それとは別に、高効率組換えDNA分子構築法の応用論文を書いているところですが詳細は秘密です。新しいヘルパープラスミドの開発は行っていません。
9.発表論文・特許などについて
この研究に関する発表論文は
Iizasa, E. and Y. Nagano, 2006
Highly efficient yeast-based in vivo DNA cloning of multiple DNA fragments
and the simultaneous construction of yeast/Escherichia coli shuttle vectors.
BioTechniques, 40, 79-83.
[PubMedへのリンク]
[発行元へのリンク](この雑誌の印刷版とオンライン版の購読(無料))(この雑誌のオンライン版の購読(無料))
です。また、この方法の植物バージョンは、
Nagano, Y., S. Takao, T. Kudo, E. Iizasa and T. Anai, 2007
Yeast-based recombineering of DNA fragments into plant transformation
vectors by one-step transformation
Plant Cell Reports, in press
DOI: 10.1007/s00299-007-0428-2.
[PubMedへのリンク]
[発行元へのリンク]
です。また、この研究に関する特許出願は、
発明者:永野幸生、飯笹英一
発明の名称:「組換えDNA分子作製法」
2005年3月国際特許出願(PCT/JP2005/005455)
です。
佐賀大に来て初めての卒論生が書いた論文が)バイオをやっている研究室ならたいてい置いてあるボチボチの雑誌に出たということで喜んでいます。(ちなみに、バイオをやっている研究室ならたいてい置いてあるのは、広告がいっぱい載っていて無料で印刷版が購読できるせいです。)
また、飯笹くんは、この研究に関する発表で、第29回蛋白質と酵素の構造と機能に関する九州シンポジウムにおいてポスター賞を受賞しました。